徠卡顯微鏡適合RNA探針
簡介
什么是RNA?
核糖核酸簡稱RNA。 這些分子是必不可少的生活的幾乎所有的工序,因為它們介導的所有步驟的基因表達的:信使RNA(mRNA)從基因轉錄,攜帶信息出細胞核。 在轉錄真核生物中,mRNA的成熟,其中涉及拆除插序列(內含子)的。 這個過程 - 被稱為拼接 - 主要是通過SN /含üRNA剪接體介導的。 在從核出口,大部分的mRNA翻譯得到的含rRNA的核糖體 - 該代碼是通過攜帶激活的氨基酸合成蛋白質轉運RNA解釋。 微RNA,短干擾RNA和的piRNA - 翻譯和特定mRNA的壽命部分由短RNA控制的。 不僅上述的RNA種類,但實際上該基因組的大部分被轉錄 - 其中的意義目前正在積極研究。
什么是顯微鏡都與RNA?
近三十年前,被發現β-actin基因分子被不均勻雞成肌細胞和成纖維細胞的細胞質中本地化。 很快,在其他模型系統 - 許多其他的例子,如芽殖酵母,果蠅和非洲爪蟾卵母細胞和哺乳動物成纖維細胞和神經元 - 緊隨其后。 最近,在果蠅胚胎和卵母細胞的RNA本地化的全基因組的研究表明,所表達的轉錄物的大部分(高達80%)分布于細胞質內的獨特的,非均勻的圖案。 此外,盡管是微小的,沒有明確定義的細胞器,細菌被證實為表達本地化的表達。
將近十年前,所推出的單分子敏感性顯微技術,它成為可以測量絕對RNA的數量 - 通過計算RNA分子 - 以前所未有的細胞或亞分辨率。 這些技術是目前允許各個單元的“深轉錄組測序”的顯微鏡載物臺的邊緣。
當然,顯微鏡,不僅提供了快照 - 位置和號碼 - RNA分子,但有合適的成像方法,轉錄深刻的機制,RNA本地化,翻譯和衰變被發現。
你怎么能想象RNA?
核糖核酸 - 類似于DNA和多肽 - 是聚合物大分子。 其身份是由主結構或積木序列中,核糖核苷酸(A,C,G和U)來決定。 大多數的RNA分子是單鏈的 - 雖然它們折疊成復雜的二級和三級結構往往涉及形成分子內雙鏈體 - 從而他們的身份可以與匹配互補序列進行探測。 這些探頭大多是單鏈自然的核酸 - 單鏈DNA或RNA - 攜帶一些檢測標記,從放射性同位素,小,免疫檢測的分子 - haptenes - 或熒光染料。 期間稱為原位雜交(ISH)的方法,該探針找到并結合其靶RNA。 不匹配的無雜交通常更穩定能量上比從而通過在一個特定信號被開發的ISH方法適當的洗滌步驟被探針形成的,其他的,非特異性復合物是。 然而,由于這些強制性分化步驟: - 在惡劣治療旁一些ISH協議要求(使用高溫,有機溶劑,高鹽等) - 沒有這些ISH技術是與生活細胞成像相容。
那你怎么跟著RNA住在哪里?
RNA永遠存在“裸”細胞內的 - 它總是在復雜的一組動態的蛋白質分子形成核糖核蛋白顆粒(的RNP)。 許多這些蛋白質分子的結合的核糖核酸直接(限制性商業慣例)。 融合這些限制性商業慣例到熒光蛋白(FPS)是一種廣泛使用的方法來可視化的RNA活。 大多數這些限制性商業慣例,在真核細胞的劇目發現 - 例如詩道芬,eIF4AIII,Hrp48A,PABP等,限制了目標特異性和任何給定的RNP只是兼職居民。 然而一些限制性商業慣例,如Pumillio同源域(PUM-HD)或CRISPR / Cas9,可以工程化以匹配給定的RNA靶。 然而,為了有效地顯現mRNPs - 通常含有單個mRNA分子 - 每個粒子幾個拷貝的RBP-FP融合體是必要的。 為了最大限度地減少每靶分子轉基因構建體的數量,在體內的RNA可視化技術的最常用的利用限制性商業慣例直系到主機。 第一他們的種類是MS2系統,由6-24份的RNA莖環結構的特定于MS2噬菌體和匹配的MS2外殼蛋白融合至FP串聯排列的。 通過引入MS2循環數組復制到目標ASH1 mRNA和封存未綁定的MCP-FP到細胞核,歌手實驗室可以想像生活中的芽殖酵母動態ASH1 mRNPs。
這些噬菌體環/環結合外殼蛋白系統的一個共同的缺點是它們依賴于RNA靶的轉基因修飾 - 大多引入另外的額外拷貝的內源性本兩個等位基因 - 和回路裝入外套陣列蛋白加入約1.5-2丙二醛到感興趣的RNP的大小。 這些變化可能會影響目標限制性商業慣例的動力(轉錄,運輸,翻譯和衰減),因此,理想情況下,所得到的結果應該與其他獨立,互補的方法進行驗證。
然后,還必須有其他的方法來遵循的RNP生活。 他們如何從以前的有什么不同?
1996年,特亞吉實驗室介紹分子信標(MBS),率先采用熒光探針。 MB都是短寡核苷酸探針用熒光團和淬滅劑對,在其兩個相對的末端。 這些末端(通常為6-7個核苷酸)是彼此互補的形成發夾結構從而定位所述熒光團旁邊的猝滅劑(暗狀態)。 于MB的目標特定中間部分(環)的雜交,所述發夾的莖被熔化,分離熒光團和淬滅劑,從而產生活化的熒光(亮狀態)。 使用的MB - 熒光探針在一般的 - 不要求區分洗滌步驟和目標的無轉基因修飾。
不幸的是,RNA / DNA雜交也不雙鏈RNA 在體內穩定。 為了解決這個問題,這些探頭的骨干進行化學修飾以隱藏細胞內核酸形成的混合體。 作為正副作用,這些修改 - phosphoribose骨干大多2'O甲基化和/或鎖核酸摻入 - 增加的混合動力車允許較短的探針的穩定性。
FIT探頭
什么是FIT代表什么?
噻唑橙(FIT)的強制插是另一種方式來獲得熒光探針。 這些探頭,由Seitz的實驗室開發,包括嵌入DNA染料,噻唑橙(TO),為堿替代 - 即更換非端子座中的寡核苷酸序列。 這種染料是一種低粘度的環境的非熒光時,例如在溶液(暗狀態)。 在探針的雜交,兩側堿基對的氫鍵大大限制了染料的扭轉運動,從而迫使它變成像狀態的插層,活化熒光(亮狀態)。 其結果是,FIT探針 - 不像的MB - 對不匹配非常敏感 - 特別是對那些在于:緊鄰。
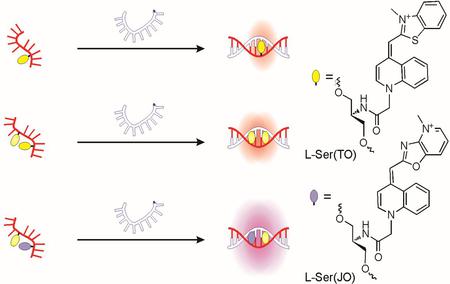
圖1:(一)單一到,(B)增加一倍,和(C)東條標記的DNA FIT-探針的示意圖。 資料來源:H?velmann等人,2013。
做這些散客探頭“樣子”?
一個熒光報道的最重要的性質是亮度。 于:(量子產率)的發射在很大程度上取決于它的微環境; 不僅在探針的雜交的狀態,而且還對位置和的鄰居核苷。 在明亮的狀態下,FIT探針雜交互補的單鏈DNA分子的典型亮度大約是7-14毫升?摩爾-1?-1,周圍20-40%的的綠色熒光蛋白和AlexaFluor 488的10-20%。然而,成像的另一個關鍵參數是此相反,即信號到其直接背景的比值。 雖然傳統的熒光團 - 像綠色熒光蛋白或AlexaFluor 488 - 都同樣明亮獨立地綁定到目標或解決方案,以黑暗狀態標記FIT探針是8-20倍,比他們的明亮狀態(響應)調光器。 此屬性使FIT探針適于例如執行原位雜交(FISH),即使在其他方式挑戰組織例如果蠅子房快速洗無熒光的。
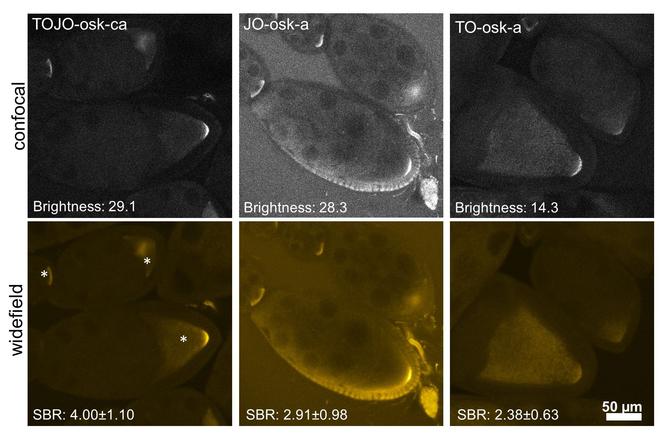
圖2: 奧斯卡 mRNA在顯影果蠅卵室清洗-自由FISH:蛋室包含顯影卵母細胞(*),可以豐富奧斯卡的mRNA最終在后極(在asterixes的右側)。 該mRNA許多人一樣是15護士細胞(左到卵母細胞)的乘積。 這16種系細胞被包封的體細胞濾泡epithelium.Source的單層:H?velmann,2013。
您可通過將多個每個探頭染料更亮FIT探頭?
不是真的 - 因為這些探針是短,典型地15-30個核苷酸長(5-10納米),兩個或更多的熒光團被定位以及熒光共振能量轉移的(FRET)的范圍內,從而淬火彼此,導致只有20亮度增加-40%。 但是,我們設法增加一倍的亮度(約24-28毫升?摩爾-1?-1)通過引入一個稍微紅移和明亮的熒光,惡唑(JO)進入到包含FIT探頭。 JO,而被嵌入染料,是相當明亮,即使在溶液中; 這是它具有低響應速度。 在單鏈探針,然而,為了有效地猝滅JO碰撞。 雜交導致亮狀態。 由于所形成的雙鏈體的剛性,TO不再猝滅JO。 相反,激發能量通過FRET傳遞從TO到JO,恢復JO的最大亮度。
另一選項以增加單個FIT探針的亮度是增加所經歷到本地約束。 我們表明,引入鎖核酸(LNA)的3',以加倍的給定探針的最大亮度,同時它離開響應有效不變。 這種效應是由于到LNA引起的減少堆垛層相鄰堿基之間的距離在雙面增加當地粘度。
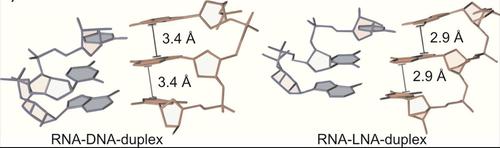
圖3:由LNA減少堆疊距離增加本地粘度。 資料來源:H?velmann和加斯帕,2014年。
你可以用這樣的亮度提高了適配性探頭的RNP體內成像?
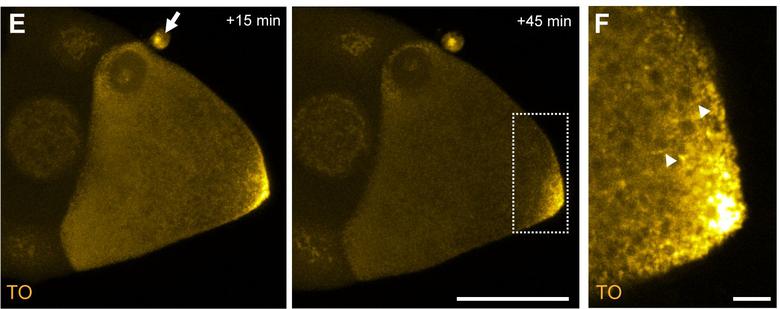
幾乎-在探頭骨干單一LNA的修改并沒有賦予足夠的核酸酶抵抗形成復式然而,與核糖(mixmer設計)的2'OH額外的甲基化,探頭被注入果蠅后保持穩定了一個多小時卵母細胞。 此外,一個探頭很少是足夠明亮,顯示個別的RNP。 通過將三至五個不同的mixmer探頭對奧斯卡的mRNA,我們設法獲得亮度和對比度相似,10倍MS2標簽標有MCP-EGFP轉基因奧斯卡的RNP。
4A
圖4:RNA體內成像:靶向奧斯卡 mRNA的3 mixmer FIT探頭的混合物注入生活,野生型卵母細胞(G,箭頭標記) 奧斯卡 RNP運動(H,箭頭)可以遵循后甚至50分鐘。注射。 ,比例尺代表50微米和5微米的G和H分別。 資料來源:H?velmann和加斯帕,2014年。
你怎么知道注射的探頭還沒有目標上的影響?
我們使用上述oskarMS2(10X)/ MCP-EGFP系統,我們和其他人有據可查,作為參考。 同時注入所有五個mixmer探測即使,我們觀察到奧斯卡 RNP活力無顯著差異相比,oskarMS2。 當這樣一個獨立的分析是不可用時,一個很好的做法可以測試多種不同的FIT探頭不同的組合。
當然,并不是所有的探頭是惰性的。 通過靶向已知雙鏈二級結構為奧斯卡 mRNA定位(一個所謂的定位元件)與mixmer探針互補的鏈(osk6)之一重要,我們誘導的奧斯卡 RNP蠕動相同的缺陷如先前的報導一系列突變奧斯卡轉基因的。 所述基于FIT探針操縱不僅少得多耗時比產生和分析的RNA的轉基因拷貝,而且只有在RNP生物合成的特定步驟的效果 - 在運輸,在這種情況下。 突變,在另一方面,可能影響從轉錄命運mRNA的開始,并且因此,所觀察到的表型可能是由于一系列的缺陷的總和。
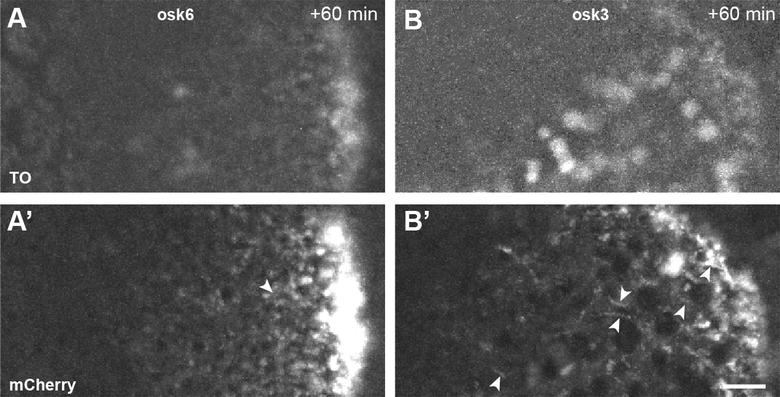
圖5:干擾使用RNP功能FIT探頭:本osk6 FIT探測目標的拼接奧斯卡本地化元素。 正如看到的TO通道(A和B)一個探頭不夠足夠明亮按照個人的RNP,但是,它會報告成功定位。 上的時間突起oskarMS2(10倍)/ MCP-mCherry(A'和B')不存在長單向奧斯卡 mRNPs(箭頭)的游程可以理解。 比例尺條為5微米。 資料來源:H?velmann和加斯帕,2014年。
總之,仔細選擇FIT探針是RNA可視化不僅是一種工具,但也RNP功能的發現。
您可以將這些探頭與其他活細胞標記,如FP標簽蛋白?
是的,可以。 由于大多數模式生物必須通過(E)綠色熒光蛋白標記的融合蛋白目前龐大的圖書館,這是至關重要的我們找到條件下,這兩個熒光團 - EGFP和TO - 可以共同觀察。 相比于:綠色熒光蛋白的吸收和發射光譜是足夠紅移,與仔細地選擇激發光 - 470毫微米為EGFP和525納米為TO - 有幾乎在一個順序掃描的兩種分子之間沒有串擾。 這些激發波長似乎有點異國風情 - 事實上,這樣的雙彩色成像,我們用一個國家的最先進的徠卡共聚焦顯微鏡配備了超連續光源。 自然,我們首先使用了oskarMS2(10倍)/ MCP-EGFP系統與FIT探針組合來檢查用于共標記和兩個信號的共遷移。
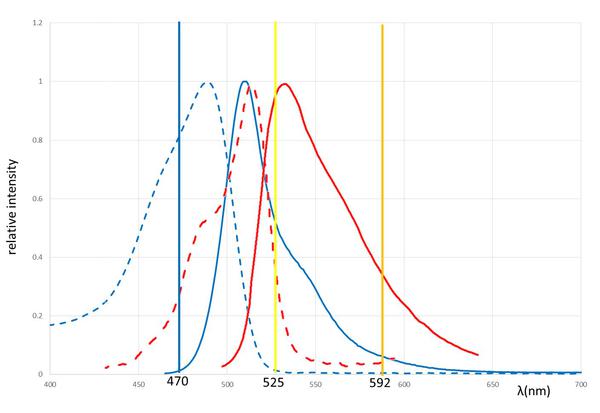
圖6:吸收和GFP的發射光譜和TO:吸收(虛線)和發射(實線)的GFP(藍色)和TO(紅色)的光譜。 的激發和耗盡激光線波長由垂直實線表示。
更有趣的應用,但是,是測試的共定位用期間的RNP的生物合成,可能有功能的蛋白質。 經典,這些發現在連接質量生化分析完成。 鏡,然而,有明顯的優點超過這樣的分析:它可以定義的地點,時間及有關的相互作用的動力學。 不幸的是,體RNP通常比衍射極限小,只包含少數 - 也許只有一個副本 - 一個給定的限制性商業慣例的。 因此,大多數免疫染色勢必造成點狀非特異性標記 - 即使特異性抗體 - 真正的RNP來自非特異性的背景難以區分。 EGFP融合蛋白的自發熒光,在另一方面,有一個幾乎百分之百的特異性和少至單個GFP分子可與當今最靈敏的檢測器來檢測。
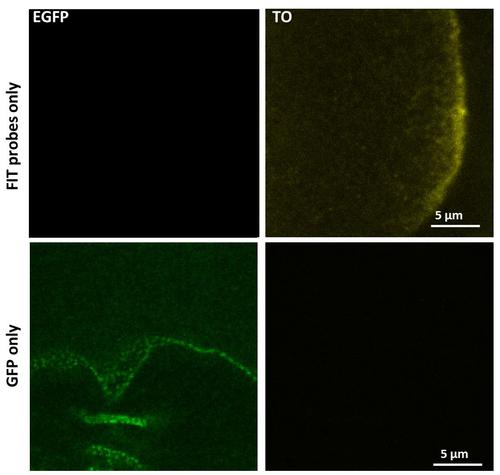
圖7:瞄準奧斯卡 mRNA和核周GFP FIT探針成像單獨使用相同的成像設置:GFP之間串擾。 有幾乎EGFP和TO之間無串擾。 資料來源:伊姆雷·加斯帕,未公布。
如果體RNP是如此之小,你有沒有考慮到的超高分辨率成像?
事實上 - 主要不是因為RNP大小,但因為擁擠,他們可以在某些位置實現。 因為果蠅卵室是一個相當厚的樣品(100微米沿軸向軸)和有趣的部分是從玻璃表面通過10-15微米厚的層細胞的分離,這限制了方法的選擇。我們發現,STED顯微鏡這些條件下工作。 LNA改性FIT探針已被證明是優良的受激發射損耗的標簽,因為在雜交時不僅它們的亮度,而且熒光壽命顯著增加,并且因此門控STED進一步提高了檢測的信號的靶特異性。 因為在EGFP和TO的發射光譜重疊的同時,一個單個592納米的激光耗竭足以實現雙色STED成像。 雖然樣本范圍最大橫向分辨率為約80-120納米,無論是個人的RNP和RNP子光散射決心做更詳細比傳統的共聚焦顯微鏡。 這種成像使我們能夠進行精確的基于對象的共定位分析。
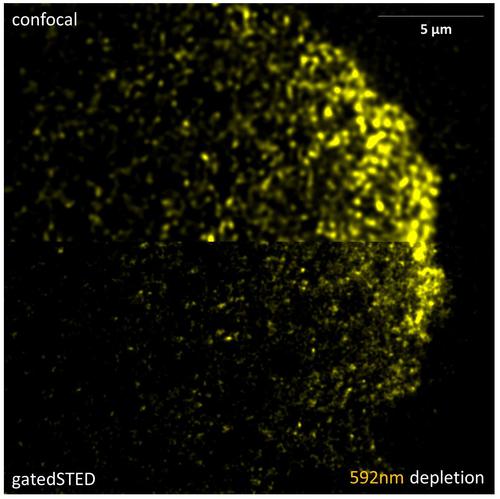
圖8:奧斯卡 mRNPs的STED成像:五LNA修飾FIT探針的混合物用于固定的果蠅卵巢。 中間oogenetic卵母細胞后極進行成像共聚焦,然后用門控STED設置。RAW圖像進行反褶積與惠更斯專業。資料來源:伊姆雷·加斯帕,未公布。
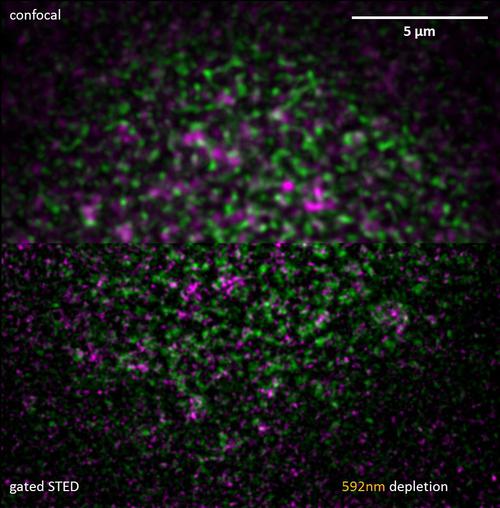
圖9:雙色STED:標有核周型綠色熒光蛋白(綠色)和瞄準奧斯卡的mRNA(品紅色)FIT探頭護士細胞的核信封。資料來源:伊姆雷·加斯帕,未公布。



